PathAI 是病理学(疾病研究)领域 AI 驱动技术工具和服务的领先提供商。我们的平台旨在通过利用机器学习的现代方法,如图像分割、图神经网络和多实例学习,大幅提高复杂疾病诊断和治疗效果测量的准确性。

传统的病理学手动分析容易出现主观性和观察者变异性,这可能对诊断和药物开发试验产生负面影响。在我们深入探讨如何使用 PyTorch 改进诊断工作流程之前,让我们首先阐述不使用机器学习的传统模拟病理学工作流程。
传统生物制药的工作方式
生物制药公司有许多途径来发现新的治疗方法或诊断方法。其中一种途径严重依赖对病理学切片的分析来回答各种问题:特定的细胞通讯途径是如何运作的?特定的疾病状态是否与特定蛋白质的存在或缺乏有关?为什么临床试验中的特定药物对某些患者有效而对另一些患者无效?患者预后与新型生物标志物之间是否存在关联?
为了帮助回答这些问题,生物制药公司依赖专业的病理学家分析切片并帮助评估他们可能遇到的问题。
正如您所想象的,需要经过专家委员会认证的病理学家才能做出准确的解释和诊断。在一项研究中,对 36 位不同的病理学家提供了单个活检结果,结果产生了 18 种不同的诊断,严重程度从无需治疗到需要积极治疗不等。在困难的边缘病例中,病理学家也经常征求同事的反馈。考虑到问题的复杂性,即使有专家培训和协作,病理学家仍然很难做出正确的诊断。这种潜在的差异可能是药物获批和临床试验失败之间的区别。
PathAI 如何利用机器学习助力药物开发
PathAI 开发机器学习模型,为药物开发研发、为临床试验提供动力以及进行诊断提供见解。为此,PathAI 利用 PyTorch 进行切片级别推理,采用多种方法,包括图神经网络 (GNN) 和多实例学习。在此背景下,“切片”指的是玻璃切片的完整扫描图像,玻璃切片是两片薄玻璃之间夹着一片薄组织,经过染色以显示各种细胞结构。PyTorch 使我们的团队能够使用这些不同的方法共享一个通用框架,该框架足够健壮,可以在我们所需的所有条件下工作。PyTorch 的高级、命令式和 Pythonic 语法使我们能够快速原型化模型,然后在获得所需结果后将这些模型投入大规模应用。
在千兆字节图像上进行多实例学习
将机器学习应用于病理学的一个独特挑战是图像的巨大尺寸。这些数字切片的分辨率通常为 100,000 x 100,000 像素或更高,大小达到千兆字节。将完整图像加载到 GPU 内存中并对其应用传统计算机视觉算法几乎是不可能完成的任务。对完整切片图像(100k x 100k)进行标注也需要大量时间和资源,特别是当标注者需要是领域专家(经委员会认证的病理学家)时。我们通常构建模型来预测患者切片上的图像级别标签,例如癌症的存在,而癌症区域在整个图像中只占据几千像素。癌变区域有时只占整个切片的很小一部分,这使得机器学习问题类似于大海捞针。另一方面,某些问题,例如预测某些组织学生物标志物,需要聚合整个切片的信息,由于图像大小的原因,这再次变得困难。所有这些因素在将机器学习技术应用于病理学问题时,都增加了显著的算法、计算和物流复杂性。
将图像分解成较小的补丁,学习补丁表示,然后汇集这些表示来预测图像级标签是解决此问题的一种方法,如下图所示。一种流行的实现方法称为多实例学习 (MIL)。每个补丁被视为一个“实例”,一组补丁构成一个“包”。各个补丁表示被汇集在一起以预测最终的包级标签。从算法上讲,包中的单个补丁实例不需要标签,因此允许我们以弱监督的方式学习包级标签。它们还使用置换不变池化函数,这使得预测与补丁的顺序无关,并允许高效地聚合信息。通常,使用基于注意力的池化函数,这不仅允许高效聚合,还为包中的每个补丁提供注意力值。这些值表示相应补丁在预测中的重要性,并且可以可视化以更好地理解模型预测。这种可解释性元素对于推动这些模型在实际世界中的采用可能非常重要,我们使用诸如加性 MIL 模型之类的变体来实现这种空间可解释性。从计算上讲,MIL 模型避开了将神经网络应用于大型图像尺寸的问题,因为补丁表示是独立于图像尺寸获得的。
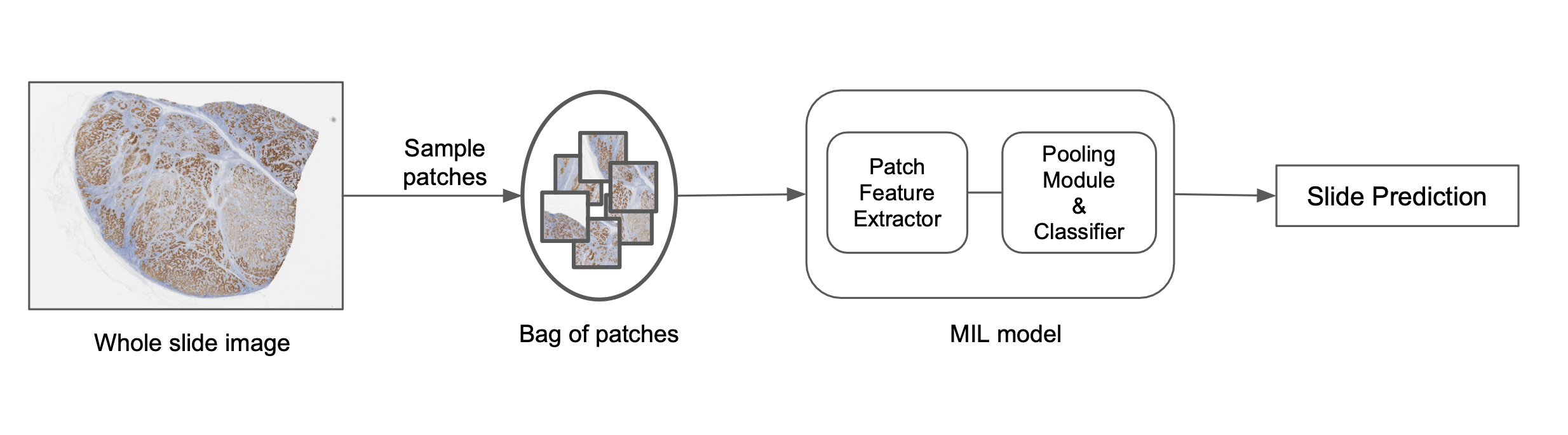
在 PathAI,我们使用基于深度网络的自定义 MIL 模型来预测图像级标签。该过程概述如下:
- 使用不同的采样方法从切片中选择补丁。
- 根据随机采样或启发式规则构建补丁包。
- 根据预训练模型或大规模表示学习模型为每个实例生成补丁表示。
- 应用置换不变池化函数以获得最终的切片级分数。
既然我们已经介绍了 PyTorch 中 MIL 的一些高级细节,那么让我们看一些代码,看看在 PyTorch 中从构思到生产代码是多么简单。我们首先定义一个采样器、变换和我们的 MIL 数据集
# Create a bag sampler which randomly samples patches from a slide
bag_sampler = RandomBagSampler(bag_size=12)
# Setup the transformations
crop_transform = FlipRotateCenterCrop(use_flips=True)
# Create the dataset which loads patches for each bag
train_dataset = MILDataset(
bag_sampler=bag_sampler,
samples_loader=sample_loader,
transform=crop_transform,
)
定义了采样器和数据集后,我们需要定义将使用该数据集进行训练的模型。PyTorch 熟悉的模型定义语法使这变得容易,同时还允许我们创建定制模型。
classifier = DefaultPooledClassifier(hidden_dims=[256, 256], input_dims=1024, output_dims=1)
pooling = DefaultAttentionModule(
input_dims=1024,
hidden_dims=[256, 256],
output_activation=StableSoftmax()
)
# Define the model which is a composition of the featurizer, pooling module and a classifier
model = DefaultMILGraph(featurizer=ShuffleNetV2(), classifier=classifier, pooling = pooling)
由于这些模型是端到端训练的,因此它们提供了一种强大的方法,可以直接从千兆像素的全切片图像生成单个标签。由于它们广泛适用于不同的生物学问题,因此其实施和部署的两个方面很重要
- 对管道的每个部分进行可配置控制,包括数据加载器、模型的模块化部分以及它们之间的交互。
- 能够快速迭代构思-实现-实验-生产循环。
PyTorch 在 MIL 建模方面具有多种优势。它提供了一种直观的方式来创建具有灵活控制流的动态计算图,这对于快速研究实验非常有用。Map-style 数据集、可配置的采样器和批量采样器允许我们自定义补丁包的构建方式,从而实现更快的实验。由于 MIL 模型是 IO 密集型模型,数据并行和 Pythonic 数据加载器使任务非常高效和用户友好。最后,PyTorch 的面向对象特性使得能够构建可重用模块,这有助于快速实验、可维护的实现以及轻松构建管道的组合组件。
使用 PyTorch 中的 GNN 探索空间组织结构
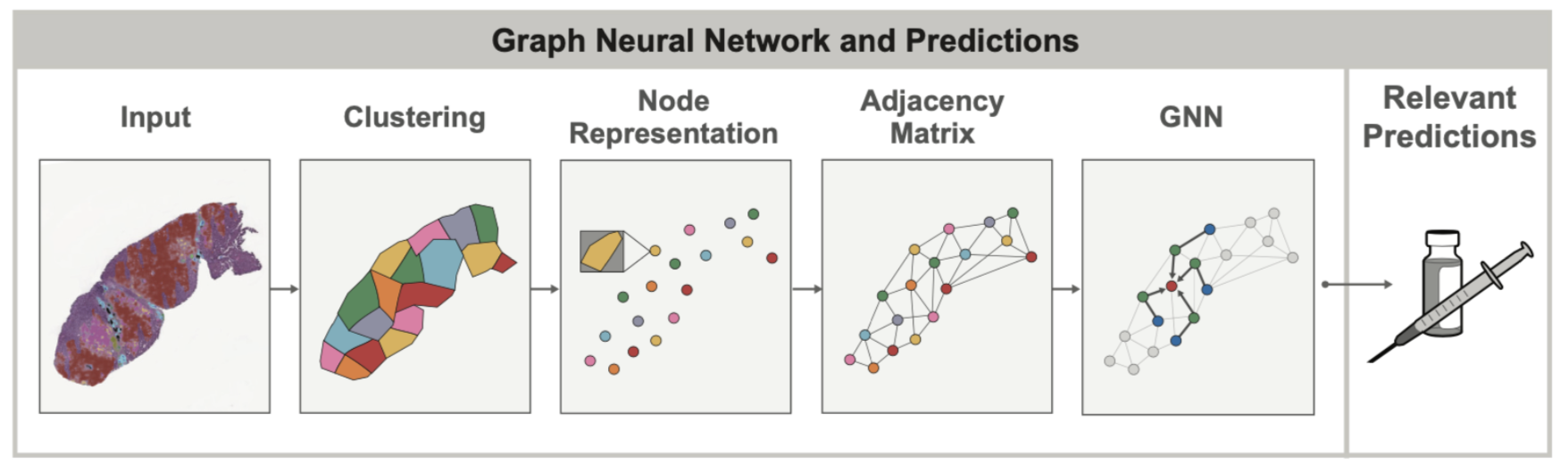
在健康和病变组织中,细胞的空间排列和结构通常与细胞本身一样重要。例如,在评估肺癌时,病理学家会尝试观察肿瘤细胞的整体分组和结构(它们是否形成实心片?还是以较小的局部簇出现?)以确定癌症是否属于具有截然不同预后的特定亚型。细胞与其他组织结构之间的这种空间关系可以使用图来建模,以同时捕获组织拓扑结构和细胞组成。图神经网络 (GNN) 允许学习这些图中与其他临床变量(例如某些癌症中基因的过度表达)相关的空间模式。
2020 年末,当 PathAI 开始在组织样本上使用 GNN 时,PyTorch 通过 PyG 包提供了最佳且最成熟的 GNN 功能支持。这使得 PyTorch 成为我们团队的自然选择,因为我们知道 GNN 模型将是我们要探索的重要 ML 概念。
GNN 在组织样本背景下的主要附加价值之一是图本身可以揭示仅凭视觉检查很难发现的空间关系。在我们最近的 AACR 出版物中,我们展示了通过使用 GNN,我们可以更好地理解肿瘤微环境中免疫细胞聚集体(特别是三级淋巴结构,或 TLS)的存在如何影响患者预后。在这种情况下,GNN 方法用于预测与 TLS 存在相关的基因表达,并识别除 TLS 区域本身之外与 TLS 相关的组织学特征。在没有机器学习模型辅助的情况下,从组织样本图像中识别此类基因表达的见解是很困难的。
我们成功应用的最有前途的 GNN 变体之一是自注意力图池化。让我们看看我们如何使用 PyTorch 和 PyG 定义自注意力图池化 (SAGPool) 模型。
class SAGPool(torch.nn.Module):
def __init__(self, ...):
super().__init__()
self.conv1 = GraphConv(in_features, hidden_features, aggr='mean')
self.convs = torch.nn.ModuleList()
self.pools = torch.nn.ModuleList()
self.convs.extend([GraphConv(hidden_features, hidden_features, aggr='mean') for i in range(num_layers - 1)])
self.pools.extend([SAGPooling(hidden_features, ratio, GNN=GraphConv, min_score=min_score) for i in range((num_layers) // 2)])
self.jump = JumpingKnowledge(mode='cat')
self.lin1 = Linear(num_layers * hidden_features, hidden_features)
self.lin2 = Linear(hidden_features, out_features)
self.out_activation = out_activation
self.dropout = dropout
在上述代码中,我们首先定义一个单一的卷积图层,然后添加两个模块列表层,这允许我们传入可变数量的层。然后我们使用空模块列表并附加可变数量的 `GraphConv` 层,接着是可变数量的 `SAGPooling` 层。我们通过添加一个跳跃知识层、两个线性层、我们的激活函数和我们的 dropout 值来完成 `SAGPool` 的定义。PyTorch 直观的语法允许我们抽象出使用 SAG Poolings 等尖端方法的复杂性,同时保持我们熟悉的模型开发的通用方法。
我们上面描述的 SAG 池模型只是 PyTorch 中的 GNN 如何让我们探索新颖想法的一个例子。我们最近还探索了多模态 CNN-GNN 混合模型,其准确性比传统病理学家共识评分高出 20%。这些创新以及传统 CNN 和 GNN 之间的相互作用再次得益于从研究到生产的短模型开发周期。
改善患者预后
为了实现我们通过 AI 驱动的病理学改善患者预后的使命,PathAI 需要依赖一个机器学习开发框架,该框架 (1) 在开发和探索的初始阶段促进快速迭代和轻松扩展(即,模型配置即代码);(2) 将模型训练和推理扩展到海量图像;(3) 轻松可靠地为我们产品的生产用途(在临床试验及其他领域)提供模型服务。正如我们所展示的,PyTorch 为我们提供了所有这些功能以及更多功能。我们对 PyTorch 的未来感到无比兴奋,迫不及待地想看看我们还能使用该框架解决哪些其他有影响力的挑战。